多细胞生物由复杂的层次性机制来维持稳态。在组织层面上,这种稳态通常由细胞内基因调控网络和细胞外环境中各种信号介导的相互作用共同维持。因此,具有空间分辨率的单细胞组学技术对理解组织微环境具有重要意义。现在国际上已有多种空间组学方法(如转录组、蛋白组)来测量单细胞级别的空间信号分布,但是仍然缺乏空间代谢组的分析方法。
SEAM方法采用高空间分辨质谱成像结合机器学习算法,实现了组织原位代谢异质性可视化、单细胞核图像识别、代谢特征信息提取以及单细胞的聚类、差异化分析,从而能够让研究者系统的解析组织中单细胞的“代谢指纹图谱(metabolic fingerprint)”(图1)。由于器官或组织中的单个细胞的代谢物图谱存在明显的空间分布的异质性,因此,定位单个细胞在组织网络中的位置、区分相关代谢物的指纹图谱差异、确定重要代谢物的分子组成有重要意义。
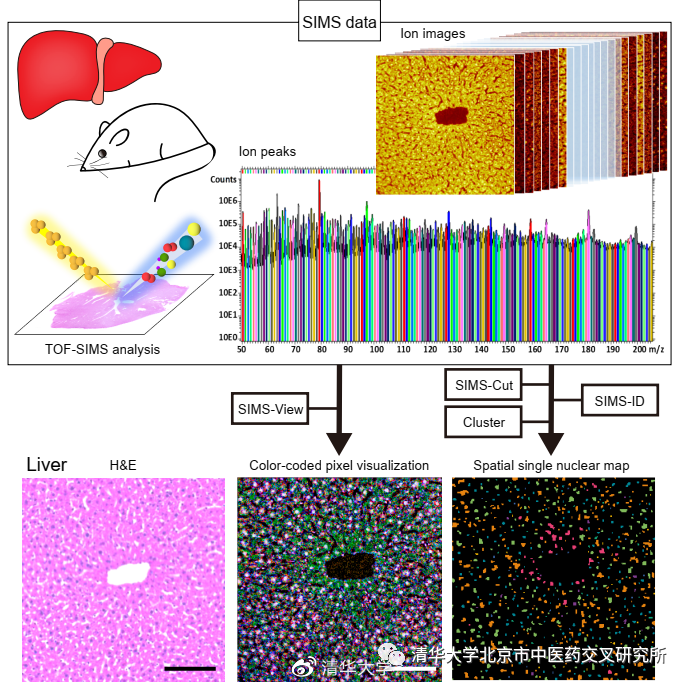
图1.SEAM在单核分辨率下捕捉到空间代谢异质性
为了验证新方法,清华大学张奇伟、张新荣团队解析了野生型小鼠肝脏组织中的空间代谢异质性,发现了肝细胞代谢异质性亚群。这一发现和前人通过蛋白或者基因表达所验证的肝组织分区现象(Liverzonation)有着高度一致(图2),有效证明了SEAM方法的可靠性和准确性。
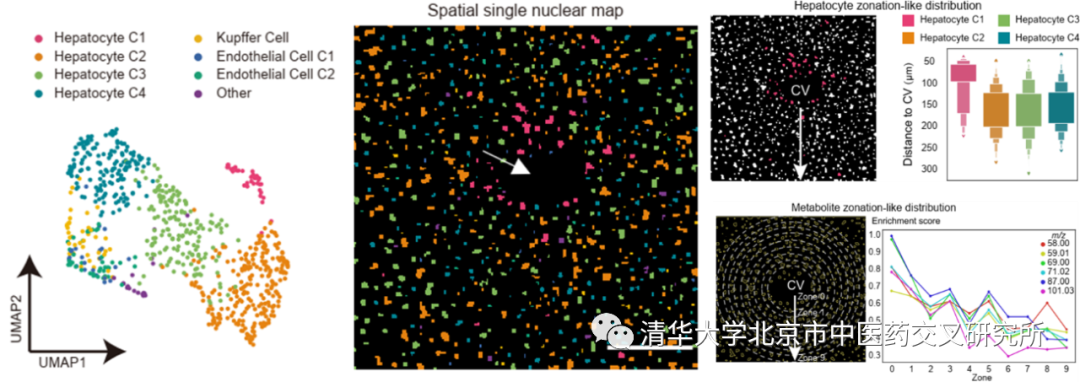
图2.SEAM在小鼠肝脏中通过代谢指纹图谱分析发现Liverzonation现象
作者还对人肝纤维化中的代谢异质性进行了空间代谢组和空间转录组联合分析。发现在肝纤维化样本中存在有两种代谢差异的肝细胞亚群,它们和纤维化区域的距离在不同样本间存在有统计学差异(图3)。通过代谢组和转录组的共同分析,发现其中的存在有谷丙酰胺的上调以及其对应代谢相关的跨膜转运蛋白的基因表达上调。虽然单细胞转录组的研究已经取得长足进展,与转录组及其表型研究密切相关的代谢组研究在单细胞水平目前还缺少方法。本文提出的“SEAM”方法,系统解析了组织空间中的单细胞代谢组,对于整个单细胞技术领域的进步具有重要的推进作用。
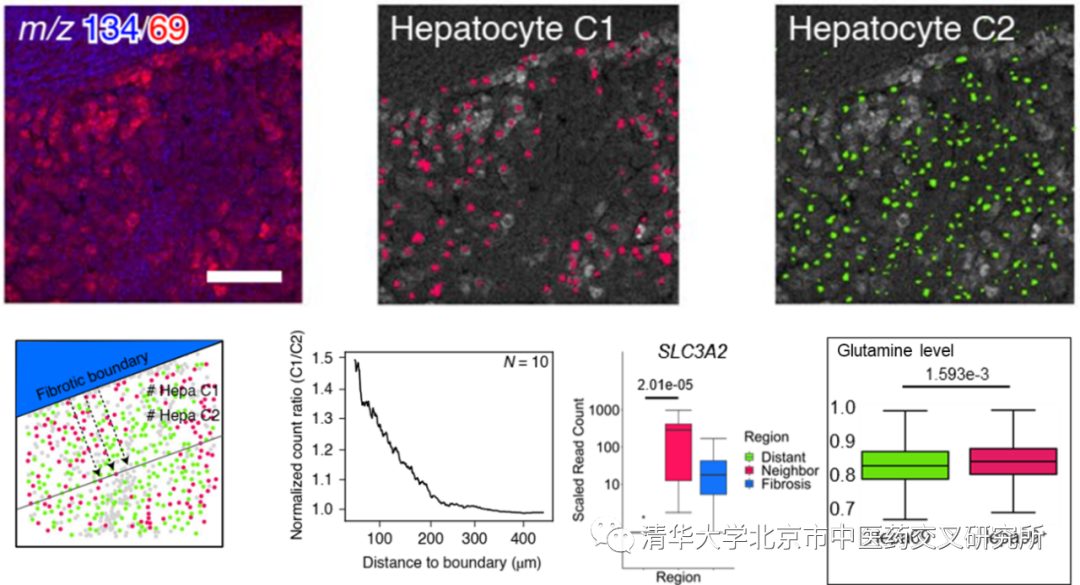
图3. SEAM在人类肝纤维化样本中发现代谢极性
相关成果以“SEAM是一个研究组织微环境的单细胞核空间代谢组学方法”(SEAM is a spatial single nuclear metabolomics method for dissecting tissue microenvironment)为题于10月4日发表于《自然·方法》(Nature Methods)。论文第一单位是清华大学自动化系。共同通讯作者是清华大学北京信息国家研究中心、医学院张奇伟教授,清华大学化学系张新荣教授,清华大学原副研究员陈阳(现中国医学科学院基础医学研究所研究员)。共同第一作者为清华大学自动化系博士生原致远、生命科学学院博士生周启明、化学系博士生蔡乐斯。中日友好医院潘林教授在本工作中参与了病理切片相关实验指导和细胞类型鉴定。北京协和医院郑永昌教授提供了临床样本。清华大学北京市中医药交叉研究所李梢教授为项目研究提出积极建议。项目由国家重点研发计划,国家自然科学基金以及北京信息国家研究中心基金等项目提供经费支持。
清华大学北京市中医药交叉研究所属于本文的第一作者单位之一。
论文链接:
https://www.nature.com/articles/s41592-021-01276-3