人体是一个由组织、细胞、分子等多层次要素构成的复杂生物系统。理解人体表型和分子的系统关联,是中西医学共同面对的一个根本问题,也是当前生物信息学、系统生物学、网络药理学等新兴学科领域关注的一个重要问题。随着目前单细胞组学等高通量检测技术的快速兴起,如何发展大数据和人工智能方法,突破跨尺度信息融合难题,解析表型与分子之间的多层次关联网络,阐释人体复杂系统运行和疾病诊疗规律,成为研究重点。近期,清华大学李梢课题组建立了基于图对比学习的人体组织-细胞-分子等多层次生物网络关系推断算法,首次构建出胃炎癌转化“组织-细胞-分子”多层次生物网络,实现了特定组织或细胞条件下疾病网络调节机理的系统分析,对于促进中西医精准诊疗具有重要意义。2022年5月,该方法以“Decoding multilevel relationships with the human tissue-cell-molecule network”为题发表于生物信息学权威刊物《Briefings in Bioinformatics》。

该文首先提出了一种基于图对比学习的多层次网络关系推断算法,实现多层次信息的系统融合以及潜在网络关联推断(图1)。该算法从国际公共数据库中系统挖掘出人体组织、细胞、分子等多层次要素的关联关系,构建出一个涵盖20,297个节点的人体“组织-细胞-分子”多层次生物网络,揭示出不同层次要素在生物网络中的模块化关联规律。在此基础上,借鉴课题组前期建立的疾病致病基因预测算法CIPHER(Molecular Systems Biology 2008, IEEE/ACM Transactions on Computational Biology and Bioinformatics 2022),提出了多层次网络关系推断算法GLIM,通过对比学习嵌入生物网络的模块化特征,实现多层次要素之间潜在网络关联的高精度推断。在多个真实数据集上的验证分析表明,GLIM算法在特定组织或细胞条件下疾病致病基因推断、细胞标志推断以及分子关联推断等方面均表现出优异的性能,精度优于同领域其他先进方法。尤为重要的是,GLIM算法还挖掘出一些新的网络关联,得到高水平文献或独立公共组学数据验证,展现出该算法在解析疾病网络调节机制方面取得新发现的潜力。
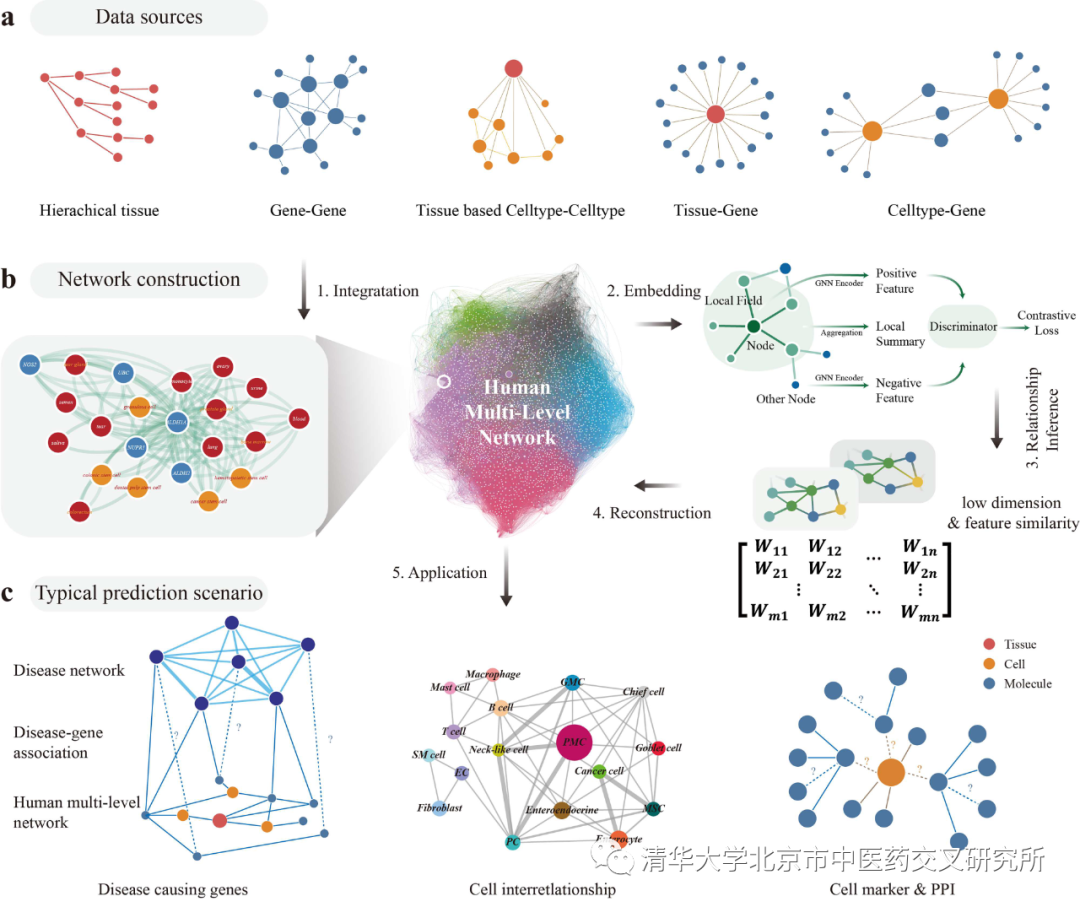
图1.解析人体多层次生物网络的GLIM算法框架
进而,以胃炎癌转化为范例,该文利用GLIM算法首次构建出胃炎癌转化“组织-细胞-分子”多层次生物网络。胃炎是胃癌发生的一个重要因素,胃炎癌转化是一个涉及组织、细胞、分子等多层次的动态过程,解析炎癌转化多层次生物网络对于理解胃癌发生机制、发掘胃癌防治标志物与干预药物具有重要的意义。李梢课题组2019年在国际上首次构建胃组织单细胞图谱,突破性发现前移胃癌早诊时点的胃癌极早期细胞,并实现临床转化应用(Cell Reports 2019)。本文将GLIM算法与胃组织单细胞图谱整合,首次构建出胃炎癌转化“组织-细胞-分子”多层次生物网络,系统解析胃炎癌转化动态过程中多层次网络节点的关联规律(图2)。通过分析不同层次网络特征,发现免疫微环境T细胞耗竭、胃粘膜上皮细胞脂肪酸和活性氧(ROS)代谢增强、非粘膜分泌上皮细胞干性相关通路活化是胃炎癌转化的关键环节,这为深入研究胃癌“极早期”新机制提供了重要切入点,也为课题组前期发现的中医寒热相关代谢-免疫网络失衡加剧促进胃炎癌转化的机制(Cancer Research 2017)提供了深入认识。
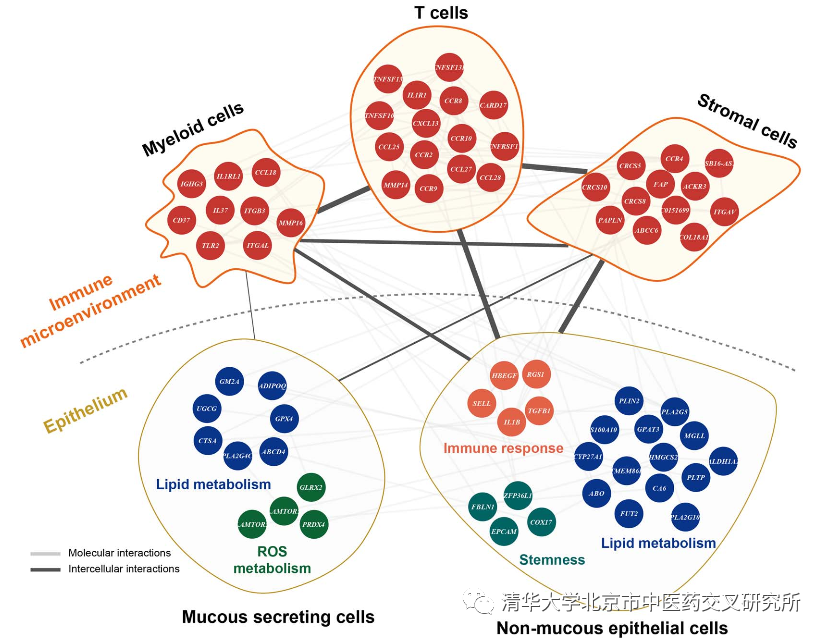
图2. 利用GLIM算法构建出的胃炎癌转化“组织-细胞-分子”多层次生物网络
李梢课题组长期致力于从生物网络角度理解中西医药的系统调节机理,提出了网络靶标理论,建立了以CIPHER算法为代表的系列高精度网络关系推断算法,开辟了中医药网络药理学方向,并在胃癌防治、中药研发等方面取得系列成果。GLIM算法将网络关系推断拓展到组织、细胞等层次,为进一步发展网络药理学、系统理解中西医药的生物基础提供了新方法。清华大学自动化系博士生侯思宇和博士后张鹏、杨扩为论文共同第一作者,李梢教授为论文通讯作者。该研究得到了国家自然科学基金委重点项目等的资助。
参考文献:
1.Hou S, Zhang P, Yang K, Wang L, Ma C, Li Y, Li S. Decoding multilevel relationships with the human tissue-cell-molecule network. Briefings in Bioinformatics 2022; bbac170. doi: 10.1093/bib/bbac170.
2.Wu X, Jiang R, Zhang M, Li S. Network-based global inference of human disease genes. Molecular Systems Biology 2008;4:189
3.Zhang P, Yang M, Zhang Y, Xiao S, Lai X, Tan A, Du S, Li S. Dissecting the single-cell transcriptome network underlying gastric premalignant lesions and early gastric cancer. Cell Reports 2019; 27,1934-1947.
4.Zhang Y, Chen L, Li S. CIPHER-SC: Disease-Gene Association Inference Using Graph Convolution on a Context-Aware Network with Single-Cell Data. IEEE/ACM Transactions on Computational Biology and Bioinformatics. 2022;19(2):819-829.
5.Guo Y, Nie Q, MacLean A, Li Y, Lei J, Li S. Multiscale modeling of inflammation-induced tumorigenesis reveals competing oncogenic and oncoprotective roles for inflammation. Cancer Research 2017;77(22):6429-6441.